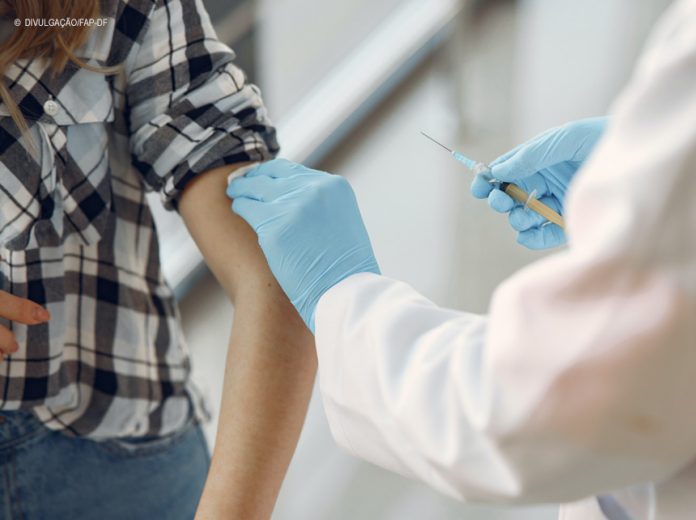
Em projeto fomentado pela Fundação de Apoio à Pesquisa e Inovação do Distrito Federal (FAP-DF), pesquisadora da Universidade de Brasília (UnB) identificou que a ocorrência de variações genéticas na bactéria causadora da coqueluche (Bordetella pertussis) podem ser responsáveis pelo aumento da incidência da doença no DF.
Principal característica da coqueluche são crises de tosse seca seguida do guincho, som produzido pelo estreitamento da glote
A coordenadora da pesquisa Identificação e vigilância das linhagens de Bordetella pertussis circulantes no Distrito Federal e Região, Tatiana Amabile de Campos, identificou que as linhagens da bactéria causadora de coqueluche circulantes no DF (sequence type fim3 e ptxP) são diferentes da linhagem utilizada nas vacinas contra a doença. “Esses dados apontam para a ocorrência de seleção vacinal, indicando a necessidade de utilização de outras linhagens para imunização”, destaca a cientista.
A coqueluche é uma infecção respiratória, transmissível, presente em todo o mundo. Sua principal característica são crises incontroláveis de tosse seca seguida pelo guincho inspiratório, que é o som produzido pelo estreitamento da glote. Segundo dados do Ministério da Saúde, em 2018, 2019 e 2020 foram confirmados respectivamente 1.165, 1.523 e 229 casos da doença no país.
Linhagens
No Distrito Federal, de acordo com Informativo Epidemiológico da Secretaria de Saúde (SES) de agosto de 2019, em 2018 foram notificados 210 casos suspeitos de coqueluche. O levantamento pode ser acessado no site da secretaria.
Para fazer a identificação e vigilância das linhagens de B. pertussis circulantes no Distrito Federal e região, entre 2007 e 2018, foi utilizada a tipagem molecular, realizada por meio da técnica de sequenciamento genético MLST/MLVA, bem como o delineamento epidemiológico das linhagens circulantes.
Durante o período do estudo, as linhagens da bactéria foram isoladas no DF e no Entorno. Esse trabalho, feito com a colaboração do Laboratório Central de Saúde do Distrito Federal (Lacen), indicou um total de 141 linhagens circulantes, até agosto de 2018.
Deste total, a cientista identificou que 94 linhagens são do tipo fim3 e ptxP; todas as linhagens são do tipo ptxP3, enquanto a linhagem utilizada na produção de vacinas é a fim3-1/ptxP2.